Перейдем к рассмотрению представленного 67-маркерного гаплотипа, и посмотрим, на какую ветвь дерева гаплотипов гаплогруппы U152 с субкладами он попадет. Это дерево приведено на рисунке, и построено из 425 гаплотипов гаплогруппы U152 в 67-маркерном формате. Представленный гаплотип находится на правой стороне дерева и помечен символом X, на него указывает угол квадратика с символом X. На дереве есть серия молодых (плоских) ветвей, но в искомой ветви таких нет, она – в старой части дерева. Общий предок всего дерева жил 3900 ± 390 лет назад, и эта датировка в точности воспроизвелась для 67– и 111-маркерных гаплотипов. Видно, как точно и надежно откалиброваны контанты скорости мутаций для 67-маркерных (0.12 мутаций на гаплотип на условное поколение) и 111-маркерных (0.198 мутаций на гаплотип на условное поколение). Число мутаций считали от базового (предкового) гаплотипа всего дерева:
13 24 14 11 11 14 12 12 12 13 13 29 – 17 9 10 11 11 25
15 19 29 15 15 16 17–11 11 19 23 16 15 18 17 36 38
12 12–11 9 15 16 8 10 10 8 10 10 12 23 23 16 10 12
12 15 8 12 22 20 13 12 11 13 11 11 12 12
Здесь отмечены 19 мутаций по отношению к представленному гаплотипу, что разводит его и базовый гаплотип дерева на 19/0.12 = 158 → 187 условных поколений, или примерно 4675 лет. Это означает, что общий предок этих двух гаплотипов, представленного и предкового, жил примерно (4675 + 3900)/2 = 4290 лет назад. Это согласуется с последними данными расчетов по геному (коллектив YFull), согласно которым субклад U152 возник между 4200 и 4900 лет назад.
Рассмотрим более внимательно ветвь, в которой находится представленный гаплотип, и которая состоит из восьми гаплотипов. На все гаплотипы приходится 139 мутаций, что дает 139/8/0.12 = 145 → 169 условных поколений, то есть 4225 ± 550 лет до общего предка. Это опять соответствует времени возникновения субкладa U152. Представленный гаплотип среди них является самым древним, потому что он отмечен терминальным снипом U152*. Итак, можно заключить, что представленный гаплотип идет напрямую от древнего общего предка, который жил примерно 4200 лет назад, и на протяжении этого времени в ДНК-линии U152* нашего коллеги не появились (или пока не обнаружены) нисходящие субклады.
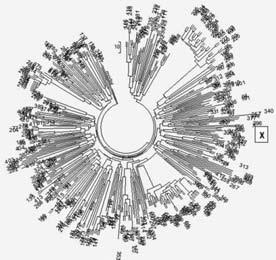
Дерево из 424 гаплотипов субклада U152 в 67-маркерном формате, к которым добавлен представленный для экспертизы гаплотип, обозначенный индексом X. Все гаплотипы дерева имеют общего предка, который жил 3900 ± 390 лет назад (та же датировка, что для 111-маркерных гаплотипов, 3900 ± 390 лет назад). На это указывают 11374 мутации, содержащиеся во всех 424 гаплотипах дерева в 111-маркерном формате (представленный гаплотип туда не входит, поскольку он 67-маркерный), что дает 11374/424/0.198 = 135 → 156 условных поколений (по 25 лет каждое), или 3900 ± 390 лет. 0.198 – константа скорости мутации для 111-маркерных гаплотипов. Для 425 гаплотипов в 67-маркерном формате (включая уже представленный гаплотип) число мутаций составляет 6867, что дает 6867/425/0.12 = 135 → 156 условных поколений, то есть ровно ту же самую величину лет до общего предка. Ветвь из 8 гаплотипов, в которую входит гаплотип X, имеет общего предка, который жил 4225 ± 550 лет назад. Гаплотипы взяты из проекта[237].
Перейдем теперь к истории субклада R1b-U152*, гаплотип которого мы рассматриваем. Древнейший этап описан в двух предыдущих историях, вплоть до прибытия эрбинов, носителей гаплогруппы R1b, на Ближний Восток. Затем носители гаплогруппы R1b-M269-L23 прошли длинным кружным путем в Европу, образуя на этом пути субклады L51 и L151, и уже на Пиренейском полуострове образовался субклад Р312, параллельно с субкладом U106. Они и составили первую волну культуры колоколовидных кубков, которая заселила континентальную Европу и Британские острова между 4800 и 3500 лет назад.
В ходе этой волны примерно 4500–4600 лет назад образовался субклад U152, дочерний по отношению к Р312. Общий предок современных носителей U152 жил примерно 3850 лет назад, и такой сдвиг общих предков к несколько (или значительно) более поздним временам случается часто, так как старые ДНК-линии отмирают, круг носителей данного субклада сужается, и формируются «новые» общие предки выжившего потомства.
В настоящее время пик распределения носителей субклада U152 (с нисходящими субкладами) приходится на Францию (в некоторых местах до 71 % мужской популяции), в ряде регионов Италии доходит до 63 % и 54 %, в Риме – 45 %, в Калабрии 44 %, в Сицилии 36 %, на Сардинии 31 %.
В данном случае наш коллега запросил, а не происходит ли он от евреев, потому что многие евреи-ашкенази относятся к гаплогруппе R1b. Хорошо, проверим этот вариант. Среди евреев носители этой гаплогруппы расходятся на несколько ДНК-линий, которые образуют «кластеры», или ветви. Одна из них, самая древняя, имеет базовый гаплотип
13 24 14 11 11 14 12 12 12 13 13 29–17 9 10 11 11 25
15 19 28 15 15 16 17–11 12 19 23 15 15 18 18 36 38
12 12
Выделены четыре мутации от базового гаплотипа U152. Общий предок этой группы евреев жил 5650 ± 710 лет назад, и четыре мутации (эквивалентные 4/0.09 = 44 → 46 условных поколений, то есть 1150 лет расхождения от общего предка U152) помещают общего предка евреев этой группы и U152 на (1150 + 5650 + 4200V2 = 5500 лет назад. Это явно предковый по отношению к U152 субклад, и может быть или L151 (расчет по снипам дает время его возникновения примерно 5600 лет назад, с коридором погрешности в интервале 5000–6300 лет назад), или L51 (время его возникновения примерно бюо лет назад, с коридором погрешности в интервале 5500–6800 лет назад, данные коллектива YFull). Иными словами, ДНК-линия евреев уходит на Ближний Восток, в шумерские времена, когда в Месопотамии мигрировали эрбины, носители гаплогруппы R1b.
Остальные ветви евреев-ашкенази гаплогруппы R1b относительно молодые, и сходятся к общим предкам, которые жили, видимо, в Европе, 1250±260, 1075±175 и 775±250 лет назад. Их базовые гаплотипы, соответственно:
12 24 14 10 11 14 11 12 12 13 14 29–17 9 10 11 11 25 15
19 30 15 15 16 16–11 10 19 23 17 16 17 17 38 38 12 12
13 24 14 11 11 14 12 12 11 13 13 28–17 9 10 11 11 26 15
19 30 15 15 15 18–11 11 19 23 16 15 18 19 36 37 12 11
12 24 14 10 11 15 12 12 12 13 14 28–15 9 9 11 11 25 14
19 29 15 15 16 17–11 12 19 23 16 17 19 17 34 38 12 12
Общий предок всех этих четырех гаплотипов ашкенази жил 5400–5600 лет назад, что полностью согласуется с приведенной выше величиной для Ближнего Востока. Таким образом, европейские евреи гаплогруппы R1b получили свои ДНК-линии не от европейцев, и не от носителей U152, в частности; это их древние линии, полученные от эрбинов, еще в доеврейские времена.
Гаплогруппа/субклад U152 пришел в Россию уже из Европы, видимо, относительно недавно, в средние века, когда в 17-м веке формировались войска иноземного строя, а затем в Россию устремились многочисленные немцы, голландцы, бельгийцы, французы, итальянцы, на государственную службу (часто получая дворянство), а также как заводчики и мелкие предприниматели. Возможно, носитель гаплотипа нашего коллеги попал в Россию именно таким путем. Об этом могут рассказать архивные материалы. К евреям он отношения не имеет.
История четвертая, родственная
Про данную историю стоит упомянуть только потому, что представленный гаплотип (в 25-маркерном формате) поступил от родного брата по отцу (то есть от дяди) героя предыдущей истории. Его гаплотип
13 25 15 11 11 11 12 12 12 12 13 28–19 9 10 11 11 25
14 19 29 15 15 16 17
в точности совпадает с первыми 25 аллелями гаплотипа племянника (см. историю третью), так что за суммарных три поколения мутации в 25-маркерном гаплотипе не произошло. Но не надо думать, что у всех племянников и их дядей гаплотипы будут всегда одинаковы, теоретическая вероятность единичного мутационного различия между ними в 25-маркерном гаплотипе равна 0.28. Это на самом деле означает, что у ста таких пар в 28 случаях мутация произойдет, а у 72 пар – не произойдет. Для 67-маркерных гаплотипов вероятность единичной мутации у дяди и племянника (или у прадедушки и правнука) равна 0.72, то есть пропорция будет уже обратной. В остальном все выводы истории предыдущей (для племянника) относятся и к его дяде.
Глава 29
Гаплогруппа S
То, что гаплогруппа S окажется у кого-то из читателей этой книги, вероятность чрезвычайно мала. Она встречается в основном среди горных папуасов, или, говоря более формально, в горных районах Папуа-Новая Гвинея, с частотой примерно 50 %, то есть в среднем у каждого второго, и в небольших количествах на соседних индонезийских островах. Изучали ее мало, и диаграмма субкладов по данным ISOGG выглядит следующим образом:

Диаграмма субкладов гаплогруппы S. Датировок образования гаплогруппы и ее субкладов коллектив YFull не приводит, их просто нет.
Глава 30
Гаплогруппа Т
Особенность гаплогруппы Т в том, что она не имеет своего «участка» на планете, где ее количество было бы существенно доминирующим по сравнению с другими гаплогруппами, хотя, похоже, это Ближний Восток. Она распределена по Земле «тонким слоем», в количествах 0.5–2%, с небольшими подъемами до 4–5% (например, в Греции, и, в частности, на Кипре, на Эгейских островах, в Сицилии, на Мальте, у сефардских евреев, у армян, азербайджанцев, лезгин, палестинцев, ливанцев, в Сирии, Саудовской Аравии, ОАЭ, Египте), но такие небольшие увеличения могут быть обусловлены просто неравномерными выборками. Даже в Саудовской Аравии, где выявлено самое высокое количество носителей гаплогруппы Т среди тестированных (в абсолютном исчислении), а именно 153 человека из 734 по всему миру в Проекте гаплогруппы Т[238](то есть 21 % от всех), относительно содержание по стране составляет всего 5 %, по сравнению с 40 % гаплогруппы J1.
В России доля гаплогруппы Т среди мужского населения[239] определена в 1.5 %. В то же время дерево гаплотипов Y-хромосомы человечества показывает, что гаплотипы гаплогруппы Т имеют того же общего предка 64 ± 6 тысяч лет назад, как и все другие неафриканские гаплотипы, и естественно вписываются в структуру дерева вместе с остальными гаплотипами. Можно предполагать, что «эпицентр» распространения гаплогруппы Т находился в регионе Ближнего Востока – восточного Средиземноморья – Кавказа, но почему он так «гладко», равномерно разошелся, остается только гадать. Впрочем, в последнее время страны Ближнего Востока стали проводить довольно массовое тестирование на гаплогруппы и субклады, и гаплогруппа Т была обнаружена практически во всех странах региона – в Ираке, Кувейте, Катаре, Бахрейне, Саудовской Аравии, а также в Судане, Египте, Италии, Испании. Из 734 членов Проекта[240] гаплогруппы Т в Саудовской Аравии, как отмечено выше, их 153 человека, в ОАЭ 56 человек, в Италии 40, в Турции 36, в Германии 35, в Кувейте 33, в Испании и Англии по 28. В России и на Украине их поровну, и суммарно 36 человек, но практически все евреи, причем в большинстве с относительно недавними временами жизни общих предков. Это будет более подробно описано ниже.
По данным коллектива YFull, гаплогруппа Т образовалась примерно 42 тысячи лет назад, с коридором погрешности плюс-минус три тысячи лет[241].
Несколько сокращенная диаграмма субкладов гаплогруппы Т приведена на рисунке ниже.

Сокращенная диаграмма субкладов гаплогруппы Т, согласно классификации ISOGG-2016. Затемнены субклады евреев в славянских странах – России, Украине, Белоруссии, Польше, Сербии, Хорватии, Чехии, Словакии, а также Литве, Латвии, Венгрии, Германии. Справа показаны времена образования некоторых субкладов по данным коллектива YFull (расчеты по снипам).
На следующем рисунке показано дерево 111-маркерных гаплотипов гаплогруппы Т.
Как обычно, дерево гаплотипов современных носителей гаплогруппы не сходится ко времени образования гаплогруппы, в данном случае примерно к 42 тысячам лет назад. Оно сходится примерно к 7200 лет назад.
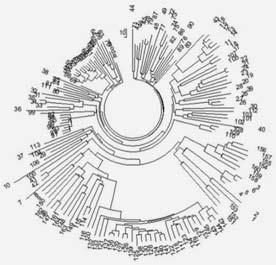
Дерево из 164 гаплотипов гаплогруппы Т в 111-маркерном формате. Все гаплотипы дерева имеют общего предка, который жил 7200 ± 725 лет назад[242] (практически та же датировка для 67-маркерных гаплотипов, 7340 ± 740 лет назад). Если вычесть плоскую относительно молодую ветвь евреев в нижней части дерева, то оставшиеся 135 гаплотипов имеют общего предка, который жил 6180 ± 620 лет назад. Гаплотипы взяты из проекта[243].
Для того чтобы получить более полное представление, кто входит в состав носителей гаплогруппы Т в России, Украине, Белоруссии, воспользуемся Проектом гаплогруппы Т, упомянутом выше. Оказалось, что все они почти исключительно евреи, причем родственные им гаплотипы, опять же евреев, выходят за пределы трех этих стран, и наблюдаются в Польше, Литве, Латвии, Чехии, Германии и других странах. Это ясно из имен и фамилий их ближайших предков (которых и заносят в базы данных). Имена такие, навскидку – Лейб, Сруль, Эфраим, Гершель, Саул, Борух, Мордехай, Шломо, Израэль, Реувен, Шимон, Янкель, Хаим, Авраам, Элиезер, Шмуль, Гершон, Моисей, Иеремия, Юфим, Бер, Саломон, Хирш, Иезекиль, Лазарь, Менахем, Ицхак, Аарон. Гаплотипы из этих стран были объединены, оставлены только те, которые имеются в 37-маркерном формате, и из них было построено дерево гаплотипов, показанное на рисунке далее.
Отсюда несколько раскрывается картина появления носителей гаплогруппы Т (и ее субкладов) в Восточной Европе. Тысячелетия назад субклады T-L131 и/или T-L208 (образовались примерно 15900 и 14800 лет назад, соответственно, по расчетам коллектива YFull) попал с Ближнего Востока к евреям или их предкам, те принесли его или нижестоящие субклады Р77 и CTS6507 во времена Рассеяния в Европу, и в интервале 800-1400 лет назад, то есть в I–II тыс нашей эры, они разошлись по Восточной Европе. Поскольку евреи обычно держались замкнуто в своих сообществах, гаплогруппа не разошлась по нееврейским жителям, и так и осталась в окружении евреев. В последняя время ситуация с замкнутостью евреев в Европе изменилась, но передаваемые гаплотипы так и остались в соответствующих ветвях. Например, гаплотип под номером 624 в древней ветви принадлежит человеку, который назвался эрзя в базе данных, а 648 – секлером из Буковины (секлеры – военное сословие в средневековой Венгрии), но их гаплотипы сидят в ветвях между евреями Украины, Белоруссии, России. Вот так раскрываются наследственные тайны, которые, как казалось в ранние времена, безвозвратно уйдут в могилу.
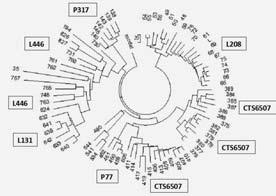
Дерево из 83 гаплотипов гаплогруппы Т стран Восточной Европы в 37-маркерном формате. На дереве видны семь относительно недавних ветвей, и одна древняя ветвь (слева). Последняя имеет общего предка, который жил 5300 ± 600 лет назад, и почти все ее гаплотипы, за исключением нескольких безымянных, принадлежат типично еврейским именам и фамилиям. Здесь представлены страны – Россия, Литва, Германия, Хорватия, Молдавия (единичный гаплотип), Венгрия, Украина, Белоруссия, Чехия. Относительно недавние ветви имеют общих предков, которые жили 790 ± 250, 910 ± 200, 1020 ± 245, 1120 ± 260, 1130 ± 190, 1175 ± 250 и 1370 ± 220 лет назад. У ветвей приведены их снипы. Гаплотипы взяты из проекта[244]
Заключение
Как гласит «Правило хорошего лектора», нужно сначала рассказать о том, о чем хочешь рассказать, потом рассказать то, что хотел рассказать, а затем рассказать, о чем рассказал.
Мы подошли к третьему этапу. Так о чем было рассказано, и к чему мы пришли? Был показан эволюционный, генеалогический путь развития современного человечества, в основном его мужской половины. Почему именно мужской, Y-хромосомной – причины довольно просты. Во-первых, эта книга о ДНК-генеалогии, а ДНК-генеалогия методологически отработана намного лучше на Y-хромосоме, мужской половой хромосоме. Для мутаций в Y-хромосоме измерены и перекрестно проверены константы скоростей мутаций, причем для каждого из ш маркеров во множестве применяемых гаплотипов. Это позволяет достаточно надежно рассчитывать хронологию древних миграций, исторических событий, изучать историю родов, из которых состоят современные популяции, этносы, нации. ДНК-генеалогия расматривает Y-хромосому как метку передвижения людей в прошлом, и это вовсе не дискриминация женщин.
Если принять крайне упрощенное представление, то мужские ДНК двигались по определенному вектору – с запада на восток, с востока на запад, с севера на юг или наоборот, хотя бы на определенных сегментах своих миграций или военных экспедиций. Это видно из рассмотрения шлейфов их древних миграций, и слово «шлейф» здесь выполняет важную смысловую функцию. Есть шлейф – есть и направление, вектор миграций, передвижений. А женщины, фигурально говоря, крутили карусель вокруг этих шлейфов, и эти карусели и обеспечивали выживание человечества, вносили разнообразие в генофонд человечества. Но вот прослеживать направление этих каруселей дело почти безнадежное. Мужские миграции были, и при этом подразумевается, что рядом с мужчинами были женщины. А вот женских миграций, наверное, не было. Именно из-за тех каруселей митохондриальных ДНК, которые на самом деле были и женскими, и мужскими, хотя бы в одном поколении.
Чтобы было понятнее – в книге приведен пример «фамильной деревни» автора, основанной почти 400 лет назад сыном боярским на земле, выданной за воинскую службу. У него была гаплогруппа R1a, и у потомков, как показали тесты на ДНК, была та же гаплогруппа. Других мужчин со стороны, «на позятьевщину», «присоседиться на невесту», в деревню не допускали, и согласно законам те, пришлые, не имели права на землю. Земля им не отписывалась. Потому что деревня была сословным статусом замкнута, изолирована, земля была выслужена, предками завещана, и нечего было пришлым там делать. Поэтому столетиями в деревне сохранялась та же гаплогруппа R1a, тот же субклад, тот же предковый гаплотип, с редкими, единичными мутациями. В деревню приходили только женщины «со стороны», как невесты и жены, и из деревни уходили в окружающие деревни только невесты и жены. Так что мтДНК крутили свою карусель через деревню столетиями, и в каждом дворе, в каждой семье была разная мтДНК. Вот так и имеем, 100 % R1a, и множество разных мтДНК в одной деревне, в одном регионе. Как здесь проследить те мтДНК как метки? Только если по материкам и континентам, «крупными мазками», в масштабе тысячелетий. Поэтому изучение мтДНК популяционными генетиками обычно приводит к конечным результатам в виде (это – реальная цитата из статьи) – «В деревне Сараево Переславского района Ярославской области выявлены следующие гаплогруппы: Н, W, I, Н, V2, Н, Н, U, X, Т*, Т*, W, Н, Н, W, T1». Понятно, что никакой ДНК-генеалогии здесь нет, и самой постановкой эксперимента не ожидалось.
Таким образом, отвечая на вопрос «что было показано» в этой книге, отметим, что была показана картина мутаций в Y-хромосоме древних предков человека на протяжении сотен тысяч лет, и показана связь этой картины мутаций с историей человечества и ветвей человечества до наших дней. Показано, как эти картины мутаций можно превратить в хронологические показатели, и фактически датировать древние и относительно недавние исторические события в годах, столетиях, тысячелетиях. Причем хронометр, позволяющий проводить датировки, не является «внешним», он встроен в наши ДНК. Поэтому в отличие, например, он «новохронологии», в которой (искусственная) датировка проводится «по понятиям», обычно приводя к совершенно абсурдным и анекдотическим результатам, расчеты в ДНК-генеалогии принципиально защищены от манипуляций «со стороны», как, например, и времена полупревращений радиоактивных элементов в физике и химии. Что ни делать, а радиоактивный распад «тикает» во времени, как ему положено по физическим законам. То же и в ДНК-генеалогии – мутации «тикают» по тем же законам, принципиальные закономерности те же, только в ДНК.
Это законы – методологическая основа ДНК-генеалогии, и она, эта основа, позволяет выстроить историю развития человечества и его ветвей на всех континентах так, как описано в этой книге.
Итак, что показано в книге – ясно. Показана ДНК-генеалогия в гаплогруппах от А до Т. Иначе говоря, ДНК-генеалогия каждого читателя, без исключения, описана в этой книге, у кого почти буквально, у кого – с высоты «птичьего полета», и чтобы она оказалась буквальной – надо просто сделать тест на гаплогруппы-субклады и гаплотипы. В этом прямую помощь окажет Академия ДНК-генеалогии, которая официально открыта в Москве в июле 2015 года (http://dna-academy.ru /), и Лаборатория ДНК-генеалогии, которая должна начать работу летом этого, 2016 года. Информационную поддержку в этом оказывает ресурс «Переформат» (http://pereformat.ru/klyosov /), на котором уже опубликованы около сотни научно-популярных статей по ДНК-генеалогии.
Для кого эта книга? Для тех, кто хочет понять историю свою и своих предков, и как эта личная история встроена в историю своего этноса, страны, всего человечества.
Как это получилось – судить читателю.
[1] Haak, W., Lazaridis, I., Patterson, N., Rohland, N., Mallick, S., Llamas, B., Brandt, G., Nordenfelt, S., Harney, E., Stewardson, K., Fu, Q., Mittnik, A., Banffy, E., Economou, C., Francken, M., Friederich, S., Pena, R.G., Hallgren, F., Khartanovich, V., Khokhlov, A., Kunst, M., Kuznetsov, P., Meller, H., Mochalov, O., Moiseyev, V., Nicklisch, N., Pichler, S.L., Risch, R., Guerra, M.A.R, Roth, C., Szecsenyi-Nagy, A., Wahl, J., Meyer, M., Krause, J., Brown, D., Anthony, D., Cooper, A., Alt, K.W., Reich, D. (2015) Massive migration from the steppe is a source for Indo-European languages in Europe. Nature 522, 207–211.
[2] Allentoft, M.E., Sikora, M., Sjogren, K.G., Rasmussen, S., Rasmussen, M., Stenderup, J., Damgaard, P.B., Schroeder, H., Ahlstrom, T., Vinner, L., Malaspinas, A.S., Margaryan, A., Higham, T., Chivall, D., Lynnerup, N., Harvig, L, Baron, J., Della Casa, P., Dąbrowski, P., Duffy, P.R., Ebel, A.V., Epimakhov, A., Frei, K., Furmanek, M., Gralak, T., et. al. (2015) Population genomics of Bronze Age Eurasia. Nature 522(7555), 167-72. doi: 10.1038/nature14507.
[3] http://www.isogg.org/tree/ISOGG_YDNATreeTrunk.html / Интернет-сайт «International Society of Genetic Genealogy».
[4] Klyosov, А.А., Rozhanskii, I.L. (2012) Re-examining the “Out of Africa” theory and the origin of Europeoids (Caucasoids) in light of DNA genealogy. Advances in Anthropology, 2, No. 2, 80–86.
[5] http://pereformat.ru/2015/10/africa-dna-vol2/
[6] Barbieri, C., H ± 1bner, A., Macholdt, Е., Ni, S., Lippold, S., Schröder, R., Mpoloka, S.W., Purps, J., Roewer, L., Stoneking, M., Pakendorf, B. (2015) Refining the Y chromosome phylogeny with southern African sequences. bioRxiv, doi: http;dx.doi.org/10.1101/034983.
[7] https://www.familytreedna.com/public/Haplogroup_A/default. aspx?section=yresults
[8] Клёсов, А.А., Килин, В.В. (2015) Калькулятор Килина-Клёсова для расчета времен до общих предков (TMRCA): новое издание. Вестник Академии ДНК-генеалогии, т. 8, № 3, стр. 321–375.
[9] Mendez, F.L., Krahn, Т., Schrack, В., Krahn, А.-М., Veeramah, K.R., Woerner, A.E., Fomine, F.L.M., Bradman, N., Thomas, M.G., Karafet, T.M., Hammer, M.F. (2013) An African American paternal lineage adds an extremely ancient root to the human у chromosome phylogenetic tree. Am. J. Hum. Genet. 92,454–459.
[10] http://www.lebialem.info/
[11] Однонули – это носители гаплогруппы A0, африканской по нынешнему месту жительства. Где эта гаплогруппа образовалась – пока неизвестно. Датировка по гаплотипам дает ее «возраст» 180 тысяч лет, датировка по снипам не дана, видимо, потому, что данных пока мало. Мы приводим это здесь только для того, чтобы показать, насколько передним краем науки это сейчас является, и как много здесь пока неясностей. Но датировка по гаплотипам 180 тысяч лет для времени образования гаплогруппы A0 более надежна, во всяком случае пока.
В любом случае, гаплогруппы A00 и A0 пока являются древнейшими, и образовались до расхождения эволюционного (Y-хромосома) дерева человечества на африканскую и неафриканскую ветви. Нам этого для концептуальных рассмотрений ДНК-генеалогии человечества пока достаточно.
Однонулей, то есть носителей гаплогруппы A0, найдено в мире всего полтора десятка человек. Они рассеяны парами и тройками по субкладам A0,
[12] https://www.familytreedna.com/public/Haplogroup_A/default. aspx?section=yresults
[13] Scozzari, R., Massaia, A., DAtanasio, Е., Myres, N.M., Perego, U.A., Trombetta, В., & Cruciani, F. (2012) Molecular dissection of the basal clades in the human Y chromosome phylogenetic tree. PLoS ONE, 7, Article ID: e49170. http://dx.doi.org/10.1371/journal
[14] Klyosov, А.А., Rozhanskii, I.L. (2012) Re-examining the “Out of Africa” theory and the origin of Europeoids (Caucasoids) in light of DNA genealogy. Advances in Anthropology, 2, No. 2, 80-86
[15] http://www.scirp.org/journal/HottestPaper.aspx?JournalID=737
[16] Klyosov, А.А., Rozhanskii, I.L. (2012) Re-examining the “Out of Africa” theory and the origin of Europeoids (Caucasoids) in light of DNA genealogy. Advances in Anthropology, 2, No. 2, 80–86.
[17] Клёсов, А.А., Килин, В.В. (2015) Калькулятор Килина-Клёсова для расчета времен до общих предков (TMRCA): новое издание. Вестник Академии ДНК-генеалогии, т. 8, № 3, стр. 321–375.
[18] http://dna-academy.ruAilin-klyosov/
[19] Klyosov, А.А., Rozhanskii, I.L. (2012) Re-examining the “Out of Africa” theory and the origin of Europeoids (Caucasoids) in light of DNA genealogy. Advances in Anthropology, 2, No. 2, 80-86
[20] Klyosov, A.A., Rozhanskii, I.L. (2012) Re-examining the “Out of Africa” theory and the origin of Europeoids (Caucasoids) in light of DNA genealogy. Advances in Anthropology, 2, No. 2, 80–86.
[21] Юрковец, В.П. (2015) Климатическая катастрофа гаплогруппы «Бета». Вестник Академии ДНК-генеалогии, т. 8, № 3, стр. 376–432.
[22] Юрковец, В.П. (2015) Климатическая катастрофа гаплогруппы «Бета». Вестник Академии ДНК-генеалогии, т. 8, № 3, стр. 376–432.
[23] Groucutt, H.S., Petraglia, M.D., Bailey, G., Scerri, E.M., Parton, A., Clark-Balzan, L., Jennings, R.P., Lewis, L., Blinkhorn, J., Drake, N.A., Breeze, P.S., Inglis, R.H., Deves, M.H., Meredith-Williams, M., Boivin, N., Thomas, M.G., Scally, A. (2015) Rethinking the dispersal of Homo sapiens our of Africa. Evolutionary Anhropology, 24,149–164.
[24] Behar, D.M., van Oven, M., Rosset, S., Metspalu, M., 100gvali, E.L., Silva, N.M., Kivisild, T., Torroni, A., Villems, R. (2012) A “Copernican” reassessment of the human mitochondrial DNA tree from its root. Amer. J. Human Genet. 90,675–684.
[25] Groucutt, H.S., Petraglia, M.D., Bailey, G., Scerri, E.M., Parton, A., Clark-Balzan, L., Jennings, R.P., Lewis, L., Blinkhorn, J., Drake, N.A., Breeze, P.S., Inglis, R.H., Deves, M.H., Meredith-Williams, M., Boivin, N., Thomas, M.G., Scally, A. (2015) Rethinking the dispersal of Homo sapiens our of Africa. Evolutionary Anhropology, 24,149–164.
[26] Behar, D.M., van Oven, М., Rosset, S., Metspalu, M., 100gvali, E.L., Silva, N.M., Kivisild, T., Torroni, A., Villems, R. (2012) A “Copernican” reassessment of the human mitochondrial DNA tree from its root. Amer. J. Human Genet. 90, 675–684.
[27] http://www.yfull.eom/tree/B/
[28] https://www.familytreedna.com/public/BYDNA/default. aspx?section=yresults
[29] Клёсов, А.А., Саидов, Х.С. (2015) Евреи и пуштуны Афганистана. Концептуал, М., стр. 441–443.
[30] http://www.isogg.org/tree/ISOGG_HapgrpB.html
[31] Barbieri, С., H ± 1bner, A., Macholdt, Е., Ni, S., Lippold, S., Schröder, R., Mpoloka, S.W., Purps, J., Roewer, L., Stoneking, M., Pakendorf, B. (2015) Refining the Y chromosome phylogeny with southern African sequences. bioRxiv, doi: http;dx.doi.org/10.1101/034983.
[32] Barbieri, С., H ± 1bner, A., Macholdt, Е., Ni, S., Lippold, S., Schröder, R., Mpoloka, S.W., Purps, J., Roewer, L., Stoneking, M., Pakendorf, B. (2015) Refining the Y chromosome phylogeny with southern African sequences. bioRxiv, doi: http;dx.doi.org/10.1101/034983.
[33] 3 3. http://www.yfull. com/tree/С/
[34] Seguin-Orlando, A., Korneliussen, T.S., Sikora, M., Malaspinas, A.-S., Manica, A., Moltke, I., A1brechtsen, A., Ко, A., Margaryan, A., Moiseyev, V., Goebel, T., Westaway, M., Lambert, D., Khartanovich, V., Wall, J.D., Nigst, P.R., Foley, R.A., Lahr, M.M., Nielsen, R., Orlando, L., Willerslev, E. (2014) Genomic structure in Europeans dating back at least 36,200 years. Supplementary Materials. Science, DOI: 10.1126/science.aaa0114
[35] Nagle, N., Ballantyne, K.N., van Oven, M., Tyler-Smith, C., Xue, Y., Taylor, D., Wilcox, S., Wilcox, L., Turkalov, R., van Oorschot, R.A., McAllister, R, Williams, L., Kayser, M., Mitchell, R J. (2015) Antiquity and diversity of aboriginal Australian Y-chromosomes. Am. J. Phys. Anthropol. doi: 10.1002/ajpa.22886, October 30, 2015
[36] Allentoft, М.Е., Sikora, М., Sjögren, K.G., Rasmussen, S., Rasmussen, M., Stenderup, J., Damgaard, P.B., Schroeder, H., Ahlström, T., Vinner, L., Malaspinas, A.S., Margaryan, A., Higham, T., Chivall, D., Lynnerup, N., Harvig, L, Baron, J., Della Casa, P., Dqbrowski, R, Duffy, P.R., Ebel, A.V., Epimakhov, A., Frei, К., Furmanek, M., Gralak, T.,et. al. (2015) Population genomics of Bronze Age Eurasia. Nature 522(7555), 167-72. doi: 10.1038/nature 14507.
[37] Olalde, I., Allentoft, M.E., Sänchez-Ouinto, F., Santpere, G., Chiang, C.W.K., DeGiorgio, M., Prado-Martinez, J… Rodriguez, J.A., Rasmussen, S., Ouilez, J., Ramirez, O., Marigorta, U.M., Fernändez-Callejo, M., Prada, M.E., Encinas, J.M.V., Nielsen, R., Netea, M.G., Novembre, J., Sturm, R.A., Sabeti, R, Marques-Bonet, T., Navarro, A., Willerslev, E., Lalueza-Fox, C. (2014) Derived immune and ancestral pigmentation alleles in a 7,000-year-old Mesolithic European. Nature 507, 225–228.
[38] Gamba, С., Jones, E.R., Teasdale, M.D., McLaughlin, R.L., Gonzalez-Fortes, G., Mattiangeli, V., Domboroczki, L., Kovari, I., Pap, I., Anders, A., Whittle, A. Dani, J., Raczky, P., Higham, T.F.G., Hofreiter, M., Bradley, D.G., Pinhasi, R. (2014) Genome flux and stasis in a five millennium transect of European prehistory. Nature Communs, DOI: 10.1038/ncomms6257
[39] https://www.familytreedna.com/public/Chaplogroup/default. aspx?section=yresults
[40] Klyosov, А.А. (2009) DNA Genealogy, mutation rates, and some historical evidences written in Y-chromosome. I. Basic principles and the method. J. Genetic Genealogy, 5,186–216.
[41] Klyosov, A. A. (2012) Ancient history of the Arbins, bearers of haplogroup R1b, from Central Asia to Europe, 16,000 to 1500 years before present. Advances in Anthropology, 2, No. 2, 87-105.
[42] Адамов, Д.С., Клёсов, А.А. (2009) Определение возраста популяций по STR гаплотипам Y-хромосомы. Часть II. Погрешности расчетов. Вестник Российской Академии ДНК-генеалогии, т. 2, № 1, стр. 93-103; Адамов, Д.С., Клёсов, А.А. (2009) Определение возраста популяций по STR гаплотипам Y-хромосомы. Часть III. Примеры «линейных» и «квадратичных» моделей с учетом степени асимметрии мутаций. Вестник Российской Академии ДНК-генеалогии, т. 2, № 2, стр. 187–199; Klyosov, А.А. (2009) DNA Genealogy, mutation rates, and some historical evidences written in Y-chromosome. I. Basic principles and the method. J. Genetic Genealogy, 5,186–216; Klyosov, A.A. (2012) Ancient history of the Arbins, bearers of haplogroup R1b, from Central Asia to Europe, 16,000 to 1500 years before present. Advances in Anthropology, 2, No. 2, 87-105
[43] Nagle, N., Ballantyne, K.N., van Oven, M., Tyler-Smith, C., Xue, Y., Taylor, D., Wilcox, S., Wilcox, L., Turkalov, R., van Oorschot, R.A., McAllister, R, Williams, L., Kayser, M., Mitchell, R J. (2015) Antiquity and diversity of aboriginal Australian Y-chromosomes. Am. J. Phys. Anthropol. doi: 10.1002/ajpa.22886, October 30, 2015
[44] https://www.familytreedna.com/public/kirgiz/default. aspx?section=yresults
[45] Zerjal, Т., Xue, Y., Bertorelle, G., Wells, R.S., Bao, W. et al. (2003) The Genetic Legacy of the Mongols. Am. J. Hum. Genet. 72, 717–721.
[46] Там же.
[47] https://www.familytreedna.com/public/Chaplogroup/default. aspx?section=yresults
[48] https://www.familytreedna.com/public/Dhaplogroup?iframe=yresults
[49] http://www.yfull. com/tree/D/
[50] 5 0. http://www.yfull. com/tree/Е/
[51] Lacan, М., Keyser, C., Ricaut, F.-X. et al. (2011) Ancient DNA suggests the leading role played by men in the Neolithic dissemination. Proc. Natl. Acad. Sei. US, 108,18255-18259.
[52] https://www.familytreedna.com/public/E3b?iframe=yresults
[53] http://www.yfull.eom/tree/E/
[54] Discussions. The mutation rate constants in DNA genealogy and related issues. Вестник Российской Академии ДНК-генеалогии, том 4, № 11, 2108–2195 (2011).
[55] https://www.familytreedna.com/public/E3b?iframe=yresults
[56] https://www.familytreedna.com/public/E-V13/default. aspx?section=yresults
[57] Haak, W., Lazaridis, I., Patterson, N., Rohland, N., Mallick, S., Llamas, В., Brandt, G., Nordenfeit, S., Harney, E., Stewardson, K., Fu, O., Mittnik, A., Bänf-fy, E., Economou, C., Francken, M., Friederich, S., Pena, R.G., Hallgren, F., Khar-tanovich, V., Khokhlov, A., Kunst, M., Kuznetsov, P., Meller, H., Mochalov, 0., Moiseyev, V., Nicklisch, N., Pichler, S.L., Risch, R., Guerra, M.A.R, Roth, C., Szec-senyi-Nagy, A., Wahl, J., Meyer, M., Krause, J., Brown, D., Anthony, D., Cooper, A., Alt, K.W., Reich, D. (2015) Massive migration from the steppe is a source for Indo-European languages in Europe. Nature 522, 207–211.
[58] Roewer, L., Willuweit, S., Kr ± 1ger, C., Nagy, M., Rychkov, S., Morozowa, L, Naumova, O., Schneider,Y., Zhukova, O., Stoneking, M., Nasidze, I. (2008) Analysis of Y chromosome STR haplotypes in the European part of Russia reveals high diversities but non-significant genetic distances between populations. Int J Legal Med., 122,219–223.
[59] Balanovsky, О., Dibirova, К., Dybo, A., Mudrak, О., Frolova, S., Pocheshkhova, Е., Haber, М., Platt, D., Schurr, T., Haak, W., Kuznetsova, M., Radzhabov, M., Balaganskaya, O., Romanov, A., Zakharova, T., Hernanz S.D.F., Zalloua, R, Koshel, S., Ruhlen, M., Renfrew. C., Wells, R.S., Tyler-Smith, C., Balanovska, E. (2011) Parallel evolution of genes and languages in the Caucasus region. Mol Biol Evol. 28, 2905–2920.
[60] Клёсов, А.А., Килин, В.В. (2015) Калькулятор Килина-Клёсова для расчета времен до общих предков (TMRCA): новое издание. Вестник Академии ДНК-генеалогии, т. 8, № 3, стр. 321–375.
[61] Клёсов, А.А., Саидов, Х.С. (2015) Евреи и пуштуны Афганистана. Концептуал, М., стр. 415–419.
[62] https://www.familytreedna.com/public/JewishDNAProject/default. aspx?section=yresults
[63] Haak, W., Balanovsky, О., Sanchez, J.J. et al. (2010) Ancient DNA from European early Neolithic farmers reveals their Near Eastern affinities. PLOS Biology, 8 (11) el000536. doi: 10.1371/journal.pbio.1000536.
[64] Клёсов, A.A. (2012) Гаплогруппы и гаплотипы Кавказа. Вестник Академии ДНК-генеалогии, том 5, № 9,1005–1036.
[65] Klyosov, А. А. (2012) Ancient history of the Arbins, bearers of haplogroup R1b, from Central Asia to Europe, 16000 to 1500 years before present. Adv. Anthropology, 2, No. 2, 87-105.
[66] Lacan, М., Keyser, С., Ricaut, F.-X. et al. (2011) Ancient DNA reveals male diffusion through the Neolithic Mediterranean route. Proc. Natl. Acad. Sei. US, 108,9788–9791.
[67] Lacan, M., Keyser, C., Ricaut, F.-X. et al. (2011) Ancient DNA suggests the leading role played by men in the Neolithic dissemination. Proc. Natl. Acad. Sei. US, 108, 18255-18259.
[68] Ermini, L., Olivieri, С., Rizzi, Е. et al. (2008) Complete mitochondrial genome sequence of the Tyrolean Iceman. Curr. Biol. 18, 1687–1693; Fu, O., Mittnik, A., Johnson, P.L.F. et al. (2013) A revised timescale for human evolution based on ancient mitochondrial genomes. Curr. Biol. 23, 1–7; Kean, W.F., Tocchio, S., Kean, M., Rainsford, K.D. (2013) The musculoskeletal abnormalities of the Similaun Iceman («ÖTZI»): clues to chronic pain and possible treatments. Inflammopharmacology, 21, 11–20; P ± 1ntener, A.G., Moss, S. (2010) Otzi, the Iceman and his leather clothes. Chimia (Aurau) 64, 315–320; Olivieri, C., Ermini, L., Rizzi, E. et al. (2010) Characterization of nucleotide misincorporation patterns in the iceman’s mitochondrial DNA. PLOS One, 5(1): e8629. doi: 10.1371/joutnal.pone.0008629; Rollo, F., Ermini, L., Luciani, S. et al. (2006) Fine characterization of the Iceman’s mtDNA haplogroup. Am. J. Phys. Anthropol. 130,557–564.
[69] https://www.familytreedna.com/public/Ossetian/default. aspx?section=yresults
[70] http://www.yfull.com/tree/G/
[71] https://www.familytreedna.com/public/KBalkarDNA/default. aspx?section=yresults
[72] Lacan, М., Keyser, С., Ricaut, F.-X., Brucato, N., Tarrus, J., Bosch, A., Guilaine, J., Crubezy, E., Ludes, B. (2011) Ancient DNA suggests the leading role played by men in the Neolithic dissemilation. Proc. Natl. Acad. Sei. US, 108(45), 18255-18259.
[73] https://www.familytreedna.com/public/Ossetian/default. aspx?section=yresults
[74] http://www.yfull. com/tree/H
[75] Sengupta, S., Zhivotovsky, L.A., King, R., Mehdi, S.O., Edmonds, C.A., Chow, C.-E.T.,Lin,A.A., Mitra, M., Sil, S.K., Ramesh, A., Rani, M.V.U.,Thakur,C.M., Cavalli-Sforza, L.L., Majumder, P.P., and Underhill, P.A. (2006) Polarity and temporality of high-resolution Y-chromosome distributions in India identify both indigenous and exogenous expansions and reveal minor genetic influence of Central Asian Pastoralis. Amer. J. Human Genet. 78, 202–221.
[76] Kivisild, Т, Rootsi, S., Metspalu, М., Mastana, S., Kaldma, К., Parik, J., Metspalu, E., Adojaan, M., Tolk, H.-V., Stepanov, V., et al. (2003) The genetic heritage of the earliest settlers persists both in Indian tribal and caste populations. Hum. Genet. 72, 313–332; Клёсов, A.A., Тюняев, A.A. (2010) Происхождение человека, Белые Альвы, стр. 315–316.
[77] Cordaux, R., Bentley, G., Aunger, G., Sirajuddin, S.M., Stoneking, M. (2004) Y-STR haplotypes from eight South Indian groups based on five loci. Forensic Sei. 49, 1–2; Клёсов, A.A., Тюняев, A.A. (2010) Происхождение человека, Белые Альвы, стр. 316–317.
[78] Клёсов, А.А., Тюняев, А.А. (2010) Происхождение человека, Белые Альвы, стр. 317–319.
[79] Клёсов, А.А., Тюняев, А.А. (2010) Происхождение человека, Белые Альвы, стр. 319–320.
[80] https://www.familytreedna.com/public/YHaploGroupH?iframe=yresults
[81] Mathieson, I., Lazaridis, I., Rohland, N., Mallick, S., Patterson, N., Roodenberg, S.A., Harney, E., Stewardson, K., Fernandes, D., Novak, M., Sirak, K., Gamba, C., Jones, E.R., Llamas, В., Dryomov, S., Pickreil, J., Arsuaga, J.L., de Castro, J.M.B., Carboneil, E., Gerritsen, F., Khokhlov, A., Kuznetsov, P., Lozano, M., Meller, Н., Mochalov, О., Moiseyev, V., Guerra, M.A.R., Roodenberg, J., Verges, J.M., Krause, J., Cooper, A., Alt, K.W., Brown, D., Anthony, D., Lalueza-Fox, C.L., Haak, W., Pinhasi, R., Reich, D. (2015) Genome-wide patterns of selection in 230 ancient Eurasians. Nature 528,499–503.
[82] Lazaridis, I., Patterson, N., Mittnik, A., Renaud, G., Mallick, S., Kirsanow, K., Sudmant, P.H., Schraiber, J.G., Castellano, S., Lipson, M., Berger, B., Economou, C., Bollongino, R., Fu, O., Bos, K.I., Nordenfeit, S., Li, H., de Filippo, C., Pr ± 1fer, K., Sawyer, S., Posth, C., Haak, W., et al. (2014) Ancient human genomes suggest three ancestral populations for present-day Europeans. Nature 513(7518) 409–413.
[83] Allentoft, M.E., Sikora, M., Sjögren, K.G., Rasmussen, S., Rasmussen, M., Stenderup, J., Damgaard, P.B., Schroeder, H., Ahlström, T., Vinner, L., Malaspinas, A.S., Margaryan, A., Higham, T., Chivall, D., Lynnerup, N., Harvig,L,Baron, J.,DellaCasa,P.,D^browski,P.,Duffy,P.R.,Ebel,A.V.,Epimakhov,A., Frei, К., Furmanek, M., Gralak, T.,et. al. (2015) Population genomics of Bronze Age Eurasia. Nature 522(7555), 167-72. doi:10.1038/naturel4507.
[84] Gamba, C., Jones, E.R., Teasdale, M.D., McLaughlin, R.L., Gonzalez-Fortes, G., Mattiangeli, V., Domboroczki, L., Kovari, L, Pap, L, Anders, A., Whittle, A. Dani, J., Raczky, P., Higham, T.F.G., Hofreiter, M., Bradley, D.G., Pinhasi, R. (2014) Genome flux and stasis in a five millennium transect of European prehistory. Nature Communs, DOI: 10.1038/ncomms6257
[85] Gamba, C., Jones, E.R., Teasdale, M.D., McLaughlin, R.L., Gonzalez-Fortes, G., Mattiangeli, V., Domboroczki, L., Kovari, L, Pap, L, Anders, A., Whittle, A. Dani, J., Raczky, P., Higham, T.F.G., Hofreiter, M., Bradley, D.G., Pinhasi, R. (2014) Genome flux and stasis in a five millennium transect of European prehistory. Nature Communs, DOI: 10.1038/ncomms6257
[86] http://www.yfull.com/tree/Il/
[87] Волков, В.Г., Каримов, А.А., Юсупов, Ю.М. (2015) Геногеография башкир рода Кыргыз гаплогруппы II. БЭИП «Суюн», 2, № 10, 972–977.
[88] http://www.eupedia.com/europe/european_y-dna_haplogroups.shtml
[89] https://www.familytreedna.com/public/yDNA_Il/default. aspx?section=yresults
[90] Клёсов, А.А. (2013) Необычные гаплотипы группы II в Башкирии и Киргизии. Вестник Академии ДНК-генеалогии, том 6, № 3, 529–531.
[91] https://www.familytreedna.com/public/I2aHapGroup/default. aspx?section=yresults
[92] Klyosov, А.А., Tomezolli, G.T. (2013) DNA genealogy and linguistics. Ancient Europe. Advances in Anthropology, 3,101–111.
[93] http://www.yfull.com/tree/I2/
[94] Lacan, М., Keyser, С., Ricaut, F.-X., Brucato, N., Duranthon, F., Guilainec, J., Crubezy, E., Ludes, B. (2011) Ancient DNA reveals male diffusion through the Neolithic Mediterranean route. Proc. Natl. Acad. Sei. US, 108, 9788–9791.
[95] Клёсов, А.А. (2012) Динарская (восточно-европейская) и «островные» ветви гаплогруппы I2а. Вестник Академии ДНК-генеалогии, том 5, № 11,1304–1317.
[96] http://www.yfull.com/tree/I2/
[97] https://www.familytreedna.com/public/HaplogroupIYDNA/default. aspx?section=yresults
[98] https://www.familytreedna.com/public/HaplogroupIYDNA/default. aspx?section=yresults
[99] http://www.yfull. com/tree/I2/
[100] https://www.familytreedna.com/public/HaplogroupIYDNA/default. aspx?section=yresults
[101] https://www.familytreedna.com/public/I2aHapGroup/default. aspx?section=yresults
[102] http://www.isogg.org/tree/ISOGG_HapgrpJ.html
[103] Szecsenyi-Nagy, А. (2015) Molecular genetic investigation of the Neolithic population history in the western Carpathian Basin. Diss. Doktor der Naturwissenschaften am Fachbereich Biologie, Gutenberg-Universität, Mainz, Germany, 356 pp.
[104] Jones, E.R., Gonzalez-Fortes, G., Connell, S., Siska, V., Eriksson, A., Martiniano, R., McLaughlin, R.L., Llorente, M.G., Cassidy, L.M., Gamba, C., Meshveliani, T., Bar-Yosef, O., Muller, W., Belfer-Cohen, A., Matskevich, Z., Jakeli, N., Higham, T.F.G., Currat, M., Lordkipanidze, D., Hofreiter, M., Manica, A., Pinhasi, R., Bradley, D.G. (2015) Upper Palaeolithic genomes reveal deep roots of modern Eurasians. Nature Communs, DOI: 10.1038/ncomms9912
[105] Gamba, C., Jones, E.R., Teasdale, M.D., McLaughlin, R.L., Gonzalez-Fortes, G., Mattiangeli, V., Domboroczki, L., Kovari, I., Pap, I., Anders, A., Whittle, A. Dani, J., Raczky, P., Higham, T.F.G., Hofreiter, M., Bradley, D.G., Pinhasi, R. (2014) Genome flux and stasis in a five millennium transect of European prehistory. Nature Communs, DOI: 10.1038/ncomms6257
[106] https://www.familytreedna.com/public/J2-M172?iframe=yresults
[107] Hammer, M.F., Behar, D.M., Karafet, Т.М., Mendez, F.L., Hallmark, В., Erez,T., Zhivotovsky, L. A., Rosset, S., Skorecki, K. (2009) Extended Y chromosome haplotypes resolve multiple and unique lineages of the Jewish priesthood. Hum. Genet. 126,707–717.
[108] Hammer, M.F., Behar, D.M., Karafet, T.M., Mendez, F.L., Hallmark, В., Erez,T., Zhivotovsky, L. A., Rosset, S., Skorecki, K. (2009) Extended Y chromosome haplotypes resolve multiple and unique lineages of the Jewish priesthood. Hum. Genet. 126,707–717.
[109] https://www.familytreedna.com/public/J-M267/default.aspx?section=yresults
[110] https://www.familytreedna.com/public/J2-M172?iframe=yresults
[111] https://www.familytreedna.com/public/J2-M172?iframe=yresults
[112] Nagle, N., Ballantyne, K.N., van Oven, M., Tyler-Smith, C., Xue, Y., Taylor, D., Wilcox, S., Wilcox, L., Turkalov, R., van Oorschot, R.A., McAllister, R, Williams, L., Kayser, M., Mitchell, R.J. (2015) Antiquity and diversity of aboriginal Australian Y-chromosomes. Am. J. Phys. Anthropol. doi: 10.1002/ajpa.22886, October 30, 2015
[113] Fu, О., Li, Н., Moorjani, R, Jay, F., Slepchenko, S.M., Bondarev, A.A., Johnson, P.L., Aximu-Petri, A., Pr ± 1fer, К., de Filippo., C, Meyer, M., Zwyns, N., Salazar-Garcia, D.C., Kuzmin, Y.V., Keates, S.G., Kosintsev, P.A., Razhev, D.I., Richards, M.P., Peristov, N.V., Lachmann, M., Douka, K., Higham, T.F., Slatkin, M., Hublin, J.J., Reich, D., Kelso, J., Viola, T.B., Pääbo, S. (2014) Genome sequence of a 45,000-year-old modern human from western Siberia. Nature. 2014 Oct 23;514(7523):445-9.
[114] http://www.yfull.eom/tree/K/
[115] http://www.yfull.eom/tree/L/
[116] https://www.familytreedna.com/public/Y-Haplogroup-L/default. aspx?section=yresults
[117] Mona, S., Tommaseo-Ponzetta, М., Brauer, S., Sudoyo, H., Marzuki, S., Kayser, M. (2007) Patterns of Y-chromosome diversity intersect with the Trans-New Guinea hypothesis. Mol. Biol. Evol., 24, 2546–2555.
[118] Nagle, N., Ballantyne, K.N., van Oven, M., Tyler-Smith, C., Xue, Y., Taylor, D., Wilcox, S., Wilcox, L., Turkalov, R., van Oorschot, R.A., McAllister, R, Williams, L., Kayser, M., Mitchell, R.J. (2015) Antiquity and diversity of aboriginal Australian Y-chromosomes. Am. J. Phys. Anthropol. doi: 10.1002/ajpa.22886, October 30, 2015
[119] Gamba, С., Jones, E.R., Teasdale, M.D., McLaughlin, R.L., Gonzalez-Fortes, G., Mattiangeli, V., Domboroczki, L., Kovari, L, Pap, I., Anders, A., Whittle, A. Dani, J., Raczky, P., Higham, T.F.G., Hofreiter, M., Bradley, D.G., Pinhasi, R. (2014) Genome flux and stasis in a five millennium transect of European prehistory. Nature Communs, DOI: 10.1038/ncomms6257
[120] Allentoft, M.E., Sikora, M., Sjögren, K.G., Rasmussen, S., Rasmussen, M., Stenderup, J., Damgaard, P.B., Schroeder, H., Ahlström, T., Vinner, L., Malaspinas, A.S., Margaryan, A., Higham, T., Chivall, D., Lynnerup, N., Harvig, L, Baron, J., Della Casa, P., D^browski, P., Duffy, P.R., Ebel, A.V., Epimakhov, A., Frei, К., Furmanek, M., Gralak, T.,et. al. (2015) Population genomics of Bronze Age Eurasia. Nature 522(7555), 167-72. doi: 10.1038/naturel4507.
[121] http://pereformat.ru/2015/12/sedov-dnk-genealogiya/
[122] http://www.eupedia.com/europe/european_y-dna_haplogroups.shtml
[123] https://www.familytreedna.com/public/N1c1/default. aspx?section=yresults
[124] https://app.box.eom/s/jo5w2jqp7bg2dksxsoc9
[125] https://www.familytreedna.com/public/N lc 1/default. aspx?section=yresults
[126] Клёсов, А.А. «Происхождение славян. ДНК-генеалогия против ‘норманнской теории’» М., 2013, 512 стр.
[127] http://www.yfull.eom/tree/N/
[128] https://www.familytreedna.com/public/o3/default. aspx?section=yresults
[129] Клёсов, А.А. (2014) Гаплогруппы и гаплотипы Синцзяня (критическое обсуждение статьи). Вестник Академии ДНК-генеалогии, том 7, № 3, 479–486.
[130] Rasmussen, М., Sikora, М., A1brechtsen, A., Korneliussen, T.S., Moreno-Mayar, J.V., Poznik, G.D., Zollikofer, C.P.E., de Leon, M.S.P., Allentoft, M.E., Moltke, L, Jonsson, H., Valdiosera, C., MalH1, R.S., Orlando, L., Bustamante, C.D., Stafford, T.W., Meitzer, D.J., Nielsen, R., Willerslev, E. (2015) The ancestry and affiliations of Kennewick Man. Nature 523,455–458.
[131] Rasmussen, М., Anzick, S.L., Waters, M.R., Skoglund, R, DeGiorgio, M., Stafford, T.W., Rasmussen, S., Moltke, I., A1brechtsen, A., Doyle, S.M., Poznik, G.D., Gudmundsdottir, V., Yadav, R., Malaspinas, A.-S., White, S.S., Allentoft, M. et al. (2014) The genome of a Late Pleistocene human from a Clovis burial site in western Montana. Nature 506, 225–229.
[132] Rasmussen, M., Li, Y., Lindgreen, S., Pedersen, J.S., A1brechtsen, A., Moltke, L, Metspalu, M., Metspalu, E., Kivisild, T., Gupta, R., Bertalan, M., Nielsen, K., Gilbert, M.T.P., Wang, Y., Raghavan, M., et al. (2010) Ancient human genome sequence of an extinct Palaeo-Eskimo. Nature 463, 757–762.
[133] Rasmussen, М., Anzick, S.L., Waters, M.R., Skoglund, R, DeGiorgio, M., Stafford, T.W., Rasmussen, S.,Moltke, I.,A1brechtsen, A., Doyle, S.M.,Poznik,G.D., Gudmundsdottir, V., Yadav, R., Malaspinas, A.-S., White, S.S., Allentoft, M. et al. (2014) The genome of a Late Pleistocene human from a Clovis burial site in western Montana. Nature 506,225–229.
[134] Raghavan, М., Skoglund, R, Graf, К.Е., Metspalu, М., A1brechtsen, А., Moltke, I., Rasmussen, S., Stafford, ТЖ, Orlando, L., Metspalu, E., Karmin, M., Tambets, K., Rootsi, S., Mägi, R., Campos, P.F. et al (2013) Upper Palaeolithic Siberian genome reveals dual ancestry of Native Americans. Nature 505, 87–91.
[135] Там же
[136] http://www.familytreedna.com/public/yDNA_Q/default. aspx?section=yresults
[137] https://www.familytreedna.com/public/Jewish_0?iframe=yresults
[138] https://www.familytreedna.com/public/Jewish_Q?iframe=yresults
[139] Baltrusch, Е. (2002) Die konstantinische lex generalis von 321 an die Stadt Köln und die Juden. In: Felten, F.J., Irrgang, S., Wesoly, K., (Eds.), Ein gef ± 1llter Willkomm: Festschrift f ± 1r Knut Schulz zum 65. Geburtstag. Shaker, Aachen, pp. 1-15.
[140] Sch ± 1tte, S., Gechter, M. (2012) Von der Ausgrabung zum Museum – Kölner Archäologie zwischen Rathaus und Praetorium; Ergebnisse und Materialien 2006–2012. B&T Bader & Team GmbH, [Vienna].
[141] Там же
[142] Там же.
[143] Klyosov, А.А. (2009) DNA Genealogy, mutation rates, and some historical evidences written in Y-chromosome. II. Walking the map. J. Genetic Genealogy, 5,217–256, и ссылки там же.
[144] Klyosov, А. А. (2012) Ancient history of the Arbins, bearers of haplogroup R1b, from Central Asia to Europe, 16,000 to 1500 years before present. Advances in Anthropology, 2, No. 2, 87-105.
[145] Raghavan, M., Skoglund, R, Graf, K.E., Metspalu, M., A1brechtsen, A., Moltke, I., Rasmussen, S., Stafford, T.W., Orlando, L., Metspalu, E., Karmin, M., Tambets, K., Rootsi, S., Mägi, R., Campos, RF. et al (2013) Upper Palaeolithic Siberian genome reveals dual ancestry of Native Americans. Nature 505, 87–91.
[146] Расчеты проведены коллективом YFull http://www.yfull.com/tree/ R1/
[147] https://www.familytreedna.com/public/R2/default. aspx?section=yresults
[148] http://www.yfull. com/tre e/R 1 а/
[149] Klyosov, А.А. (2009) DNA Genealogy, mutation rates, and some historical evidences written in Y-chromosome. II. Walking the map. J. Genetic Genealogy, 5,217–256, и ссылки там же.
[150] Haak, W., Brandt, G., de Jong, H.N. et al. (2008) Ancient DNA, strontium isotopes, and osteological analyses shed light on social and kinship organization of the later Stone Age. Proc. Natl. Acad. Sei. US, 105,18226-18231.
[151] Клёсов, A.A. (2008) Откуда появились славяне и «индоевропейцы»? Ответ дает ДНК-генеалогия. Вестник Российской Академии ДНК-генеалогии, том 1, № 3, август 2008, стр. 400–478.
[152] Haak, W., Brandt, G., de Jong, H.N. et al. (2008) Ancient DNA, strontium isotopes, and osteological analyses shed light on social and kinship organization of the later Stone Age. Proc. Natl. Acad. Sei. US, 105,18226-18231.
[153] Haak, W., Lazaridis, L, Patterson, N., Rohland, N., Mallick, S., Llamas, В., Brandt, G., Nordenfeit, S., Harney, E., Stewardson, K., Fu, O., Mittnik, A., Bänffy, E., Economou, C., Francken, M., Friederich, S., Pena, R.G., Hallgren, F., Khartanovich, V., Khokhlov, A., Kunst, M., Kuznetsov, P., Meller, H., Mochalov, O., Moiseyev, V., Nicklisch, N., Pichler, S.L., Risch, R., Guerra, M.A.R, Roth, C., Szecsenyi-Nagy, A., Wahl, J., Meyer, M., Krause, J., Brown, D., Anthony, D., Cooper, A., Alt, K.W., Reich, D. (2015) Massive migration from the steppe is a source for Indo-European languages in Europe. Nature 522, 207-211.
[154] Szecsenyi-Nagy, А. (2015) Molecular genetic investigation of the Neolithic population history in the western Carpathian Basin. Diss. Doktor der Naturwissenschaften am Fachbereich Biologie, Gutenberg-Universität, Mainz, Germany, 356 pp.
[155] http://elementy.ru/novosti_nauki/432506/Paleogenetika_podtverdila vazhnyy_vklad_prichernomorsko_kaspiyskikh_stepnyakov_v_formirovanie genofondaevropeytsev
[156] Haak, W., Lazaridis, I., Patterson, N., Rohland, N., Mallick, S., Llamas, В., Brandt, G., Nordenfeit, S., Harney, E., Stewardson, K., Fu, O., Mittnik, A., Bänffy, E., Economou, C., Francken, M., Friederich, S., Pena, R.G., Hallgren, F., Khartanovich, V., Khokhlov, A., Kunst, M., Kuznetsov, P., Meller, H., Mochalov, O., Moiseyev, V., Nicklisch, N., Pichler, S.L., Risch, R., Guerra, M.A.R, Roth, C., Szec-senyi-Nagy, A., Wahl, J., Meyer, M., Krause, J., Brown, D., Anthony, D., Cooper, A., Alt, K.W., Reich, D. (2015) Massive migration from the steppe is a source for Indo-European languages in Europe. Nature 522, 207–211.
[157] Allentoft, M.E., Sikora, M., Sjögren, K.G., Rasmussen, S., Rasmussen, M., Stenderup, J., Damgaard, P.B., Schroeder, H., Ahlström, T., Vinner, L., Malaspinas, A.S., Margaryan, A., Higham, T., Chivall, D., Lynnerup, N., Harvig, L, Baron, J., Della Casa, P., D^browski, R, Duffy, P.R., Ebel, A.V., Epimakhov, A., Frei, К., Furmanek, M., Gralak, T.,et. al. (2015) Population genomics of Bronze Age Eurasia. Nature 522(7555), 167-72. doi: 10.1038/naturel4507.
[158] Klyosov, А.А. (2012) Ancient history of the Arbins, bearers of haplo-group R1b, from Central Asia to Europe, 16,000 to 1500 years before present. Advances in Anthropology, 2, No. 2, 87-105.
[159] Чекунова, Е.М., Ярцева, Н.В., Чекунов, М.К., Мазуркевич, А.Н. (2014) Первые результаты генотипирования коренных жителей и человеческих костных останков из археологических памятников верхнего Подвинья. Археология озерных поселений IV–II тыс до н. э. Материалы международной конференции, посвященной полувековому исследованию свайных поселений на северо-западе России. Санкт-Петербург, 13–15 ноября 2014 г., стр. 287–294.
[160] Там же
[161] Там же
[162] Haak, W., Lazaridis, I., Patterson, N., Rohland, N., Mallick, S., Llamas, В., Brandt, G., Nordenfeit, S., Harney, E., Stewardson, K., Fu, O., Mittnik, A., Bänffy, E., Economou, C., Francken, M., Friederich, S., Pena, R.G., Hallgren, F., Khartanovich, V., Khokhlov, A., Kunst, M., Kuznetsov, P., Meller, H., Mochalov, O., Moiseyev, V., Nicklisch, N., Pichler, S.L., Risch, R., Guerra, M.A.R, Roth, C., Szec-senyi-Nagy, A., Wahl,Meyer, M., Krause, J., Brown, D., Anthony, D., Cooper, A., Alt, K.W., Reich, D. (2015) Massive migration from the steppe is a source for Indo-European languages in Europe. Nature 522, 207–211.
[163] Allentoft, M.E., Sikora, M., Sjögren, K.G., Rasmussen, S., Rasmussen, M., Stenderup, J., Damgaard, P.B., Schroeder, H., Ahlström, T., Vinner, L., Malaspinas, A.S., Margaryan, A., Higham, T., Chivall, D., Lynnerup, N., Harvig, L, Baron, J., Della Casa, P., D^browski, R, Duffy, P.R., Ebel, A.V., Epimakhov, A., Frei, К., Furmanek, M., Gralak, T.,et. al. (2015) Population genomics of Bronze Age Eurasia. Nature 522(7555), 167-72. doi: 10.1038/naturel4507.
[164] Keyser, С., Bouakaze, С., Crubezy, Е., Nokolaev, V.G., Montagnon, D., Reis, T., Ludes, В. (2009) Ancient DNA provides new insights into the history of south Siberian Kurgan people. Human Genetics 126, 395–410.
[165] Haak, W., Lazaridis, L, Patterson, N., Rohland, N., Mallick, S., Llamas, В., Brandt, G., Nordenfeit, S., Harney, E., Stewardson, K., Fu, O., Mittnik, A., Bänffy, E., Economou, C., Francken, M., Friederich, S., Pena, R.G., Hallgren, F., Khartanovich, V., Khokhlov, A., Kunst, M., Kuznetsov, P., Meller, H., Mochalov, O., Moiseyev, V., Nicklisch, N., Pichler, S.L., Risch, R., Guerra, M.A.R, Roth, C., Szecsenyi-Nagy, A., Wahl, J., Meyer, M., Krause, J., Brown, D., Anthony, D., Cooper, A., Alt, K.W., Reich, D. (2015) Massive migration from the steppe is a source for Indo-European languages in Europe. Nature 522, 207–211.
[166] Keyser, С., Bouakaze, С., Crubezy, Е. et al. (2009) Ancient DNA provides new insight into the history of south Siberian Kurgan people. Hum. Genet. 126, 395–410.
[167] Allentoft, М.Е., Sikora, М., Sjögren, K.G., Rasmussen, S., Rasmussen, M., Stenderup, J., Damgaard, P.B., Schroeder, H., Ahlström, T., Vinner, L., Malaspinas, A.S., Margaryan, A., Higham, T., Chivall, D., Lynnerup, N., Harvig, L, Baron, J., Della Casa, R, D^browski, R, Duffy, RR., Ebel, A.V., Epimakhov, A., Frei, К., Furmanek, M., Gralak, T.,et. al. (2015) Population genomics of Bronze Age Eurasia. Nature 522(7555), 167-72. doi: 10.1038/naturel4507.
[168] Allentoft, М.Е., Sikora, М., Sjögren, K.G., Rasmussen, S., Rasmussen, M., Stenderup, J., Damgaard, P.B., Schroeder, H., Ahlström, T., Vinner, L., Malaspinas, A.S., Margaryan, A., Higham, T., Chivall, D., Lynnerup, N., Harvig, L, Baron, J., Della Casa, R, D^browski, R, Duffy, RR., Ebel, A.V., Epimakhov, A., Frei, К., Furmanek, M., Gralak, T.,et. al. (2015) Population genomics of Bronze Age Eurasia. Nature 522(7555), 167-72. doi: 10.1038/naturel4507.
[169] Allentoft, М.Е., Sikora, М., Sjögren, K.G., Rasmussen, S., Rasmussen, M., Stenderup, J., Damgaard, P.B., Schroeder, H., Ahlström, T., Vinner, L., Malaspinas, A.S., Margaryan, A., Higham, T., Chivall, D., Lynnerup, N., Harvig, L, Baron, J., Della Casa, R, D^browski, R, Duffy, RR., Ebel, A.V., Epimakhov, A., Frei, К., Furmanek, M., Gralak, T.,et. al. (2015) Population genomics of Bronze Age Eurasia. Nature 522(7555), 167-72. doi: 10.1038/naturel4507.
[170] Li, С., Li, LL, Cui, Y. et al. (2010) Evidence that a West-East admixed population lived in the Tarim Basin as early as the early Bronze Age. BMC Biology, 8:15, dohlO.l 186/1741-7007-8-15.
[171] Li, С., Li, LL, Cui, Y. et al. (2010) Evidence that a West-East admixed population lived in the Tarim Basin as early as the early Bronze Age. BMC Biology, 8:15, doi:10.1186/1741-7007-8-15.
[172] Abu-Amero, К.К., Hellani, А., Gonzalez, А.М., Larruga, J.M., Cabrera, V.M., Underhill, P.A. (2009) Saudi Arabian Y-chromosome diversity and its relationship with nearby regions. BMC Genet. 10, 59.
[173] Клёсов, A.A. (2015) Славяне, кавказцы, евреи с точки зрения ДНК-генеалогии. М., Книжный мир, 351 стр.
[174] Behar, D.M., Thomas, M.G., Skorecki, К., Hammer, M.F., Bulygina, E., Rosengarten, D., Jones, A.L., Held, K., Moses, V., Goldstein, D., Bradman, N and Weale, M.E. Multiple origins of Ashkenazi Levites: Y chromosome evidence for both Near Eastern and European ancestries. Am. J. Hum. Genet. 73, 768 – 779 (2003).
[175] Rootsi, S., Behar, D.M., Jarve, M., Lin, A.A., Myres, N.M., Passarelli, B., Poznik, G.D., Tzur, S., Sahakyan, H., Pathak, A.K., Rosset, S., Metspalu, M., Grug-ni, V., Semino, O., Metspalu, E., Bustamante, C.D., Skorecki, K., Villems, R., Kivisild, Т., Underhill, P.A. (2013) Phylogenetic applications of whole Y-chro-mosome sequences and the Near Eastern origin of Ashkenazi Levites. Nature Communications, DOI: 10.1038/ncomms3928
[176] Atzmon, G., Hao, Li., Pe’er, I., Velez, C., Pearlman, A., Palamara, P.F., Morrow, B., Friedman, E., Oddoux, C., Burns, E., Ostrer, H. et al. (2010) Abraham’s children in the genome era: major Jewish diaspora populations comprise distinct genetic clusters with shared Middle Eastern Ancestry. Am. J. Hum. Genet. 86, 850–859.
[177] Rozhanskii, I.L., Klyosov, А.А. (2012) Haplogroup R1a, its subclades and branches in Europe during the last 9,000 years. Advances in Anthropology, 2, No. 3,139–156.
[178] В.Стасов. Происхожденiе русскихъ былинъ. Вѣстник Европы. Журнал истории, политики, литературы. Петербург, 1868, стр. 169–221. Harvard College Library.
[179] https://www.familytreedna.com/public/India/default.aspx?section=yresults
[180] https://www.familytreedna.com/public/India/default.
aspx?section=yresults
[181] Underhill, Р.А., Poznik, G.D., Rootsi, S., Jarve, M., Lin, A.A., Wang, J., Passarelli, B., Kanbar, J., Myres, N.M., King, RJ., Cristofaro, J.D., Sahakyan, H., Behar, D.M., Kushniarevich, A., Sarac, J.S., Saric, T., Rudan, P., Pathak, A.K., Chaubey, G., Grugni, V., Semino, O., Yepiskoposyan, L., Bahmanimehr, A., Farjadian, S., Balanovsky, O., Khusnutdinova, E.K., Herrera, R.J., Chiaroni, J., Bustamante, C.D., Ouake, S.R., Kivisild, T., Villems, R. (2015) The phylogenetic and geographic structure of Y-chromosome haplogroup Rla. European Journal of Human Genetics 23,124–131.
[182] Там же.
[183] Клёсов, А.А., Саидов, Х.С. Евреи и пуштуны Афганистана. Пропавшие колена израилевы: история, политика, ДНК-генеалогия. М., Концепту-ал, 2015,460 стр.
[184] Там же
[185] https://www.familytreedna.com/public/kirgiz/default. aspx?section=yresults
[186] https://www.familytreedna.com/public/alash/default. aspx?section=yresults
[187] Муратов, Б.А. (2016) ДНК-генеалогия татарских фамилий. 2. Крымские татары. БЭИП «Суюн», 3, № 1, 28–70.
[188] http://www.eupedia.com/europe/european_y-dna_haplogroups.shtml
[189] Клёсов А.А. (2015) Венеты и венеды по данным ДНК-генеалогии. Исторический формат, No. 2, с. 75–102.
[190] Haak, W., Lazaridis, I., Patterson, N., Rohland, N., Mallick, S., Llamas, В., Brandt, G., Nordenfeit, S., Harney, E., Stewardson, K., Fu, O., Mittnik, A., Bänffy, E., Economou, C., Francken, M., Friederich, S., Pena, R.G., Hallgren, F., Khartanovich, V., Khokhlov, A., Kunst, M., Kuznetsov, P., Meller, H., Mochalov, O., Moiseyev, V., Nicklisch, N., Pichler, S.L., Risch, R., Guerra, M.A.R, Roth, C., Szec-senyi-Nagy, A., Wahl, J., Meyer, M., Krause, J., Brown, D., Anthony, D., Cooper, A., Alt, K.W., Reich, D. (2015) Massive migration from the steppe is a source for Indo-European languages in Europe. Nature 522, 207–211.
[191] А.А. Клёсов (2013) Дети боярские, или История одного русского рода, http://pereformat.ru/2013/12/deti-boyarskie/
[192] А.А. Клёсов. Интернет. Заметки научного сотрудника. М., изд. Московского университета, 2010, стр. 417–451.
[193] http://pereformat.ru/2015/03/veneti-2/
[194] Клёсов А.А. (2015) Венеты и венеды по данным ДНК-генеалогии. Исторический формат, No. 2, с. 75–102.
[195] Klyosov, А.А. (2009) DNA Genealogy, mutation rates, and some historical evidences written in Y-chromosome. II. Walking the map. J. Genetic Genealogy, 5,217–256.
[196] Klyosov, A. A. (2012) Ancient history of the Arbins, bearers of haplogroup R1b, from Central Asia to Europe, 16,000 to 1500 years before present. Advances in Anthropology, 2, No. 2, 87-105.
[197] Balter, М. (2013) Ancient DNA links native Americans with Europe. Science, 342,409–410.
[198] Raghavan, M., Skoglund, R, Graf, K.E., Metspalu, M., A1brechts-en, A., Moltke, I., Rasmussen, S., Stafford Jr. T.W., Orlando, L., Metspalu, E. et al. (2013) Upper Palaeolithic Siberian genome reveals dual ancestry of Native Americans. Nature, doi:10.1038/naturel2736
[199] Haak, W., Lazaridis, I., Patterson, N., Rohland, N., Mallick, S., Llamas, В., Brandt, G., Nordenfeit, S., Harney, E., Stewardson, K., Fu, O., Mittnik, A., Bänf-fy, E., Economou, C., Francken, M., Friederich, S., Pena, R.G., Hallgren, F., Khar-tanovich, V., Khokhlov, A., Kunst, M., Kuznetsov, P., Meller, H., Mochalov, 0., Moiseyev, V., Nicklisch, N., Pichler, S.L., Risch, R., Guerra, M.A.R, Roth, C., Szec-senyi-Nagy, A., Wahl, J., Meyer, M., Krause, J., Brown, D., Anthony, D., Cooper, A., Alt, K.W., Reich, D. (2015) Massive migration from the steppe is a source for Indo-European languages in Europe. Nature 522, 207–211.
[200] Lashgary, Z., Khodadadi, A., Singh, Y., Houshmand, S.M., Mahjoubi, F., Sharma, R, Singh, S., Seyedin, M., Srivastava, A., Ataee, M., Mohammadi, Z.S., Rezaei, N., Bamezai, R.N., Sanati, M.H. (2011) Y chromosome diversity among the Iranian religious groups: a reservoir of genetic variation. Ann Hum Biol 38 (3),364–371.
[201] Klyosov, А.А. (2012) Ancient history of the Arbins, bearers of haplo-group R1b, from Central Asia to Europe, 16,000 to 1500 years before present. Advances in Anthropology, 2, No. 2, 87-105
[202] А.А. Клёсов (2010) Основная загадка во взаимоотношениях индоевропейской и тюркской языковых семей и попытка ее решения с помощью ДНК-генеалогии: соображения нелингвиста. Вестник Российской Академии ДНК-генеалогии, т. 3, № 1,2-65.
[203] А.А. Клёсов (2015) Славяне, кавказцы, евреи с точки зрения ДНК-генеалогии. Глава «Откуда взялись «новые европейцы»? М., Книжный мир, стр.260–350.
[204] Клёсов, А.А. (2008) Загадки «западноевропейской» гаплогруппы R1b. Вестник Российской Академии ДНК-генеалогии, т. 1, № 4, 568–629.
[205] Klyosov, А.А. (2012) Ancient history of the Arbins, bearers of haplo-group R1b, from Central Asia to Europe, 16,000 to 1500 years before present. Advances in Anthropology, 2, No. 2, 87-105.
[206] Клёсов, A.A. (2010) Гаплогруппа R1b (часть 1). Вестник Российской Академии ДНК-генеалогии, т. 3, № 2, 249–299.
[207] Klyosov, А.А. (2012) Ancient history of the Arbins, bearers of haplo-group R1b, from Central Asia to Europe, 16,000 to 1500 years before present. Advances in Anthropology, 2, No. 2, 87-105.
[208] Клёсов, A.A. (2010) Гаплогруппа R1b (часть 1). Вестник Российской Академии ДНК-генеалогии, т. 3, № 2, 249–299.
[209] Sharma, S., Rai, Е., Sharma, R, Jena, M., Singh, S., DarvisH1, K., Bhat, A.K., Bhanwer, AJS, Tiwari, RK., Bamezai, R.N.K. (2009) The Indian origin of paternal haplogroup R1al* substantiates the autochthonous origin of Brahmins and the caste system. J. Human Genetics, 54,47–55.
[210] А.А. Клёсов (2015) Славяне, кавказцы, евреи с точки зрения ДНК-генеалогии. Глава «Откуда взялись «новые европейцы»? М., Книжный мир, стр.260–350.
[211] Клёсов А.А. (2015) Читая В.В. Седова «Происхождение и ранняя история славян» с точки зрения ДНК-генеалогии. Исторический формат, No. 4, с. 174–228
[212] Haak, W., Lazaridis, I., Patterson, N., Rohland, N., Mallick, S., Llamas, В., Brandt, G., Nordenfeit, S., Harney, E., Stewardson, K., Fu, O., Mittnik, A., Bänf-fy, E., Economou, C., Francken, M., Friederich, S., Pena, R.G., Hallgren, F., Khar-tanovich, V., Khokhlov, A., Kunst, M., Kuznetsov, P., Meller, H., Mochalov, O., Moiseyev, V., Nicklisch, N., Pichler, S.L., Risch, R., Guerra, M.A.R, Roth, C., Szec-senyi-Nagy, A., Wahl,Meyer, M., Krause, J., Brown, D., Anthony, D., Cooper, A., Alt, K.W., Reich, D. (2015) Massive migration from the steppe is a source for Indo-European languages in Europe. Nature 522, 207–211.
[213] Allentoft, M.E., Sikora, M., Sjögren, K.G., Rasmussen, S., Rasmussen, M., Stenderup, J., Damgaard, P.B., Schroeder, H., Ahlström, T., Vinner, L., Malaspinas, A.S., Margaryan, A., Higham, T., Chivall, D., Lynnerup, N., Harvig, L, Baron, J., Della Casa, P., D^browski, R, Duffy, P.R., Ebel, A.V., Epimakhov, A., Frei, К., Furmanek, M., Gralak, T.,et. al. (2015) Population genomics of Bronze Age Eurasia. Nature 522(7555), 167-72. doi: 10.1038/naturel4507.
[214] Allentoft, М.Е., Sikora, М., Sjögren, K.G., Rasmussen, S., Rasmussen, M., Stenderup, J., Damgaard, P.B., Schroeder, H., Ahlström, T., Vinner, L., Malaspinas, A.S., Margaryan, A., Higham, T., Chivall, D., Lynnerup, N., Harvig, L, Baron, J., Della Casa, R, D^browski, R, Duffy, RR., Ebel, A.V., Epimakhov, A., Frei, К., Furmanek, M., Gralak, T.,et. al. (2015) Population genomics of Bronze Age Eurasia. Nature 522(7555), 167-72. doi: 10.1038/naturel4507.
[215] http://pereformat.ru/2014/05/arbins-2
[216] Haak, W., Lazaridis, I., Patterson, N., Rohland, N., Mallick, S., Llamas, В., Brandt, G., Nordenfeit, S., Harney, E., Stewardson, K., Fu, O., Mittnik, A., Bänf-fy, E., Economou, C., Francken, M., Friederich, S., Pena, R.G., Hallgren, F., Khar-tanovich, V., Khokhlov, A., Kunst, M., Kuznetsov, P., Meller, H., Mochalov, 0., Moiseyev, V., Nicklisch, N., Pichler, S.L., Risch, R., Guerra, M.A.R, Roth, C., Szec-senyi-Nagy, A., Wahl, J., Meyer, M., Krause, J., Brown, D., Anthony, D., Cooper, A., Alt, K.W., Reich, D. (2015) Massive migration from the steppe is a source for Indo-European languages in Europe. Nature 522, 207–211.
[217] Allentoft, М.Е., Sikora, М., Sjögren, K.G., Rasmussen, S., Rasmussen, M., Stenderup, J., Damgaard, P.B., Schroeder, H., Ahlström, T., Vinner, L., Malaspinas, A.S., Margaryan, A., Higham, T., Chivall, D., Lynnerup, N., Harvig, L, Baron, J., Della Casa, P., D^browski, R, Duffy, P.R., Ebel, A.V., Epimakhov, A., Frei, К., Furmanek, M., Gralak, T.,et. al. (2015) Population genomics of Bronze Age Eurasia. Nature 522(7555), 167-72. doi: 10.1038/naturel4507.
[218] Haak, W., Lazaridis, I., Patterson, N., Rohland, N., Mallick, S., Llamas, B., Brandt, G., Nordenfeit, S., Harney, E., Stewardson, K., Fu, O., Mittnik, A., Bänf-fy, E., Economou, C., Francken, M., Friederich, S., Pena, R.G., Hallgren, F., Khar-tanovich, V., Khokhlov, A., Kunst, M., Kuznetsov, P., Meller, H., Mochalov, 0., Moiseyev, V., Nicklisch, N., Pichler, S.L., Risch, R., Guerra, M.A.R, Roth, C., Szec-senyi-Nagy, A., Wahl, J., Meyer, M., Krause, J., Brown, D., Anthony, D., Cooper, A., Alt, K.W., Reich, D. (2015) Massive migration from the steppe is a source for Indo-European languages in Europe. Nature 522, 207–211.
[219] Martiniano, R., Cafell, А., Holst, М., Hunter-Mann, К., Montgomery, J., Muldner, G., McLaughlin, R., Tesdale, M.D. et al. (2016) Genomic signals of migration and continuity in Britain before the Anglo-Saxons. Nature Communications, doi:10.1038/ncommsl0326, с дополнительной расшифровкой Richard Rocca, частное сообщение
[220] А. Лобов (2009) дисс. канд. биол. наук, Уфа.
[221] Там же.
[222] https://www.familytreedna.com/public/Bashqort_Tribes_and_Clans/ default.aspx?section=yresults
[223] Sharma, S., Rai, Е., Sharma, R, Jena, M., Singh, S., DarvisH1, K., Bhat, A.K., Bhanwer, AJS, Tiwari, P.K., Bamezai, R.N.K. (2009) The Indian origin of paternal haplogroup R1al* substantiates the autochthonous origin of Brahmins and the caste system. J. Human Genetics, 54,47–55.
[224] А.А. Клёсов (2014) Гаплогруппы и гаплотипы уйгуров Синцзяня. Вестник Академии ДНК-генеалогии, т. 7, № 3,479–486.
[225] Shan, W., Ablimit, A., Zhou, W., Zhang, F., Ma, Z., Zheng, X. (2014) Genetic polymorphism of 17 Y chromosomal STRs in Kazakh and Uighur populations from Xinjiang, China. Int. J. Legal Med., January 2014.
[226] Klyosov, А.А. (2014) A comment on the Paper: Were the First Europeans Pale or Dark Skinned? Advances in Anthropology, 2014,4, pp. 222–226.
[227] Thomas Krahn, частное сообщение
[228] Klyosov, A.A. (2012) Ancient history of the Arbins, bearers of haplogroup R1b, from Central Asia to Europe, 16,000 to 1500 years before present. Advances in Anthropology, 2, No. 2, 87-105.
[229] http://www.yfull. com/tre e/R 1b/
[230] http://www.yfull. com/tre e/R 1 b/
[231] http://www.eupedia.com/europe/Haplogroup_Rlb_Y-DNA.shtml
[232] http://www.eupedia.com/europe/Haplogroup_Rlb_Y-DNA.shtml
[233] http://www.eupedia.com/europe/Haplogroup_R1b_Y-DNA.shtml
[234] https://www.familytreedna.com/public/ht35new?iframe=yresults
[235] https://www.familytreedna.com/public/ht35new?iframe=yresults
[236] https://www.familytreedna.com/public/U106?iframe=yresults
[237] https://www.familytreedna.com/public/R1b-U152?iframe=yresults
[238] https://www.familytreedna.com/public/Y-Haplogroup-K2/default. aspx?section=yresults
[239] http://www.eupedia.com/europe/european_y-dna_haplogroups.shtml
[240] https://www.familytreedna.com/public/Y-Haplogroup-K2/default. aspx?section=yresults
[241] http://www.yfull.eom/tree/T/
[242] Клёсов, А.А. (2015) Калькулятор Килина-Клёсова для расчета времен до общих предков (TMRCA): новое издание. Вестник Академии ДНК-генеалогии, том 8, № 3, 321–375.
[243] https://www.familytreedna.com/public/Y-Haplogroup-K2/default. aspx?section=yresults
[244] https://www.familytreedna.com/public/Y-Haplogroup-K2/default. aspx?section=yresults
Дата: 2019-03-05, просмотров: 323.